На чем основана информационная емкость днк
Надёжное хранение информации в ДНК (2,2 петабайта на грамм)
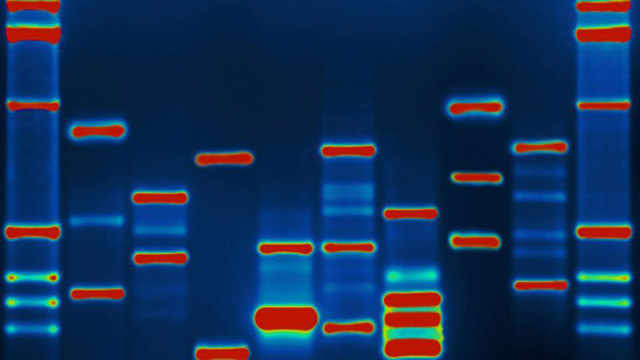
На Хабре неоднократно упоминались экспериментальные технологии записи/считывания информации в ДНК. Молекула ДНК хранит информацию в четверичной системе счисления, по количеству нуклеотидов (0 = A, 1 = T, 2 = C, 3 = G). Это компактный контейнер с плотностью записи в тысячи раз больше, чем у существующих носителей. Однако, чтобы технология перешла от научных испытаний к коммерческому использованию, требуется решить ряд проблем. Одна из них — специфика цифровой информации, в которой одни и те же биты могут многократно повторяться (CCCCCCCCCCCCCCC). Если многократно повторять один и тот же нуклеотид в молекуле ДНК, то это негативно влияет на стабильность кластера и информация может быть потеряна, даже при использовании избыточного дублирования и коррекции ошибок.
Исследователи из Европейского института биоинформатики опубликовали работу с описанием способа, как можно существенно повысить стабильность ДНК. Попросту, они предлагают отказаться от четверичной системы (Base-4) в пользу троичной (Base-3), а четвёртый нуклеотид использовать в служебных целях для разбиения длинных цепочек (CCCACCCACCCACCCACCC).
При переходе с Base-4 на Base-3 мы теряем 25% информационной ёмкости, но даже в таком варианте учёные сообщают об информационной плотности записи 2,2 петабайта на 1 грамм биологического материала. Эксперимент показал надёжность считывания информации 100%. Теоретически, эта схема способна масштабироваться в пределах, превышающих объёмы всей существующей цифровой информации, пишут авторы исследования.
Исходя из нынешнего технологического прогресса в области синтеза и секвенирования, носители ДНК для записи информации должны появиться в открытой продаже в течение десяти лет. Хотя ДНК позволяет хранить информацию тысячелетиями, первые коммерческие носители будут продаваться с гарантией до 50-ти лет, считают исследователи.
ДНК: идеальный накопитель
Вопреки распространённому мнению, недолговечность — отнюдь не главная проблема придуманных человеком цифровых накопителей информации. В самом деле, перфокарты и перфоленты способны сохранять данные на протяжении сотен лет, но часто ли вы использовали их в последние сорок? Магнитные барабаны, ленты и диски гарантируют десятки лет беспроблемного хранения — и тем не менее в самых современных персоналках их тоже уже не видно. Не успев осыпаться, выходят из моды оптические диски, и даже NAND эволюционирует на глазах — и лет через десять (как раз номинальный срок для таких чипов) прочитать сегодняшние «флэшки» наверняка будет проблематично. Кто-то грезит «голографией», но, положа руку на сердце, верите ли вы, что она протянет дольше своих предшественников?
Правильный ответ таким образом заключается не в собственно сроке жизни, а в быстрой смене технологий хранения информации. С одной стороны, этот технологический галоп — благо, ибо позволяет нам наращивать информационные аппетиты, не задумываясь об ограничениях. С другой, это же и проблема: ведь даже мелочь вроде семейного фотоальбома чёрт знает на что записать, чтобы десятилетием позже его по-прежнему можно было просмотреть! С массивами специальных данных ситуация куда хуже: «ключи» к форматам и интерфейсам быстро теряются, после чего про информацию проще забыть.
Потратив полвека на разработку идеального накопителя, мы должны признать, что идеал по-прежнему не достигнут. Каждый год приносит что-нибудь новенькое, что-нибудь более быстрое или ёмкое. И может быть поэтому всё чаще слышится голос небольшой группы энтузиастов-отщепенцев, призывающих почти буквально «вернуться к истокам». Их ругают, называя их труды бесполезной тратой времени, но за последние пятнадцать лет они добились бесспорных успехов. Здесь «группа» не означает, что люди работают в стенах одного института или компании, а только лишь, что объединёны общей идеей. И идея в целом простая: идеальный накопитель информации давно построен. Природой. Это спираль молекулы ДНК.
Несколько упрощая, можно сказать, что для живой клетки спираль ДНК выступает в качестве программы. Клетка, словно станок с числовым программным управлением, считывает с ленты ДНК инструкции, следуя которым, собирает белок. Информационная плотность её огромна: согласно последним оценкам, в одном грамме ДНК можно записать свыше 400 эксабайт — и чтобы представить, насколько велика эта цифра, вспомните, что ещё несколько лет назад всю информацию, накопленную человечеством, оценивали менее чем в две тысячи эксабайт. Даже сделав скидку на неточность, это позволяет считать спираль ДНК самым плотным способом упаковки цифровых данных из всех, практически доступных человеку.
Уникальную вместимость ДНК легко объяснить, воспользовавшись бритвой Оккама: природа, пытаясь минимизировать неблагоприятные внешние воздействия, очевидно, решила задачу хранения информации применением наименьшего количества материалов. Впрочем, собственно ёмкость — опять-таки не самое ценное, что есть в ДНК. Куда ценнее её «технологическая» зрелость.
Считается, что механизм хранения генетической информации появился на Земле одновременно с самой жизнью, то есть как минимум 3,5 миллиарда лет назад (была то спираль ДНК или родственная ей спираль РНК, в контексте нашего разговора несущественно). К сожалению, до нас не дошли столь древние ДНК-цепочки (в естественной среде такая молекула существует около миллиона лет), но те фрагменты, что удалось выделить из законсервированных в кристаллах соли образцов четвертьмиллиардной давности, имеют ту же структуру, что и ДНК-спираль в современных живых существах. Мы можем прочитать их, используя те же инструменты, которыми пользуемся для чтения ДНК дрозофилы, свиньи, человека. Иначе говоря, эффективность хранения информации в ДНК удостоверена самим временем: ни один из придуманных нами накопителей и близко не стоит по степени отлаженности.
Прежде всего, спираль ДНК не может быть бесконечно длинной — отчасти из-за естественных ограничений, отчасти по причине несовершенства современной «пишущей» техники. Это вынуждает разбивать «файл» на отрезки, вмещающие примерно сто байт каждый. Для чтения, впрочем, ДНК всё равно тоже пришлось бы резать, поэтому основная проблема — пометить отрезки, чтобы после чтения их можно было собрать в первоначальном порядке. Принимая во внимание механическую, термическую, химическую хрупкость ДНК (отклонение от комфортных для белковой жизни условий сказывается на сохранности нуклеотидных цепочек губительно), необходимо принять меры и для коррекции ошибок. Всё это заставляет помечать отрезки порядковыми номерами и подвергать помехоустойчивому кодированию (например, кодом Рида-Соломона — тем самым, который применяется, в частности, для оптических дисков).
Последнее достижение, о котором сейчас трубит научная пресса, связано как раз с защитой от неблагоприятных воздействий и принадлежит швейцарским учёным. Они научились обёртывать отрезки ДНК-спирали в нанометровых размеров защитную оболочку из кварцевого стекла. Таким образом удалось, сохранив на ДНК рекордные 83 килобайта данных, прочитать их спустя неделю хранения при температуре 70 градусов Цельсия. При отрицательных температурах данные на таком накопителе сохранились бы на протяжении миллионов лет.
При всей сенсационности, эта работа даёт представление о том, как далеки мы от использования ДНК в качестве искусственного накопителя. Писать в ДНК — всё ещё очень медленное и дорогое удовольствие: запись вышеупомянутых восьми десятков килобайт встала в полторы тысячи американских долларов. Читать из ДНК — удовольствие столь же медленное и дорогое: чтение человеческого генома (длина которого, грубо, измеряется в гигабайтах) хотя бы за тысячу долларов пока остаётся мечтой. К тому же читать можно только файл целиком, доступ к произвольной ячейке организовать удастся лишь после того, как мы научимся читать ДНК единой лентой, без разделения на фрагменты. Но по крайней мере ДНК легко копируется, а это в свою очередь позволяет оптимистам рисовать следующую заманчивую картинку недалёкого будущего.
Итак, представьте, что себестоимость записи и чтения спирали ДНК упала на несколько порядков (к тому идёт). Узнать собственный геном и определить свою предрасположенность к опасным заболеваниям теперь — дело пяти минут и одного визита к врачу. Но теперь грех не применить ДНК и в качестве идеального накопителя. Файл X записывается на ДНК-спираль и (в биореакторе размером со среднюю кастрюлю) тиражируется миллионами миллионов копий. Каждая из них обёртывается защитной оболочкой (хотя бы и по методу, предложенному швейцарцами), после чего помещается в некую инертную жидкость, сливается в пробирку и отправляется, например, в морозильную камеру, где сможет пролежать сотни лет. Или в подземное хранилище на полюсах, где пролежит миллионы.
Не слишком практично? Не слишком. Семейный фотоархив так хранить вряд ли кто-нибудь станет. Однако ёмкость и гарантированная технологическая зрелость такого накопителя позволяют решить с его помощью задачу куда более масштабную и, в общем, важную для цивилизации: организовать архив всей накопленной человеком информации.
ДНК-архив способен вместить всё, вплоть до самых мелких личных страничек и мгновенных сообщений, практически каждый бит, сгенерированный человечеством. Всё то, что влияет на нас, но нигде не сохраняется, и без чего восстановить ход событий для потомков будет невозможно (эту проблему называют цифровой амнезией). Гарантом сохранности станет сама природа: механизм записи и чтения спирали ДНК не изменился за миллиарды лет — значит, хватит и на наш век!
Геном как хранилище информации: как и зачем искусственно сохранять данные в ДНК
В цепочку генов можно записать в 60 раз больше информации, чем на сегодняшние носители. Теперь можно копировать информацию с любого цифрового носителя напрямую в ДНК, фактически превращая клетки живых организмов в миниатюрные устройства для записи и хранения данных. Рассказываем, как это работает.
Читайте «Хайтек» в
Какая информация есть в ДНК
ДНК представляет собой последовательность нуклеотидов. Их всего четыре: аденин, гуанин, тимин, цитозин.
Для кодирования информации каждому из них приписывают цифру-код. Например, тимин — 0, гуанин — 1, аденин — 2, цитозин — 3.
Последовательность нуклеотидов позволяет «кодировать» информацию о различных типах РНК. Все эти типы РНК синтезируются на матрице ДНК за счет копирования последовательности ДНК в последовательность РНК, синтезируемой в процессе транскрипции, и принимают участие в биосинтезе белков (процессе трансляции).
Помимо кодирующих последовательностей, ДНК клеток содержит последовательности, выполняющие регуляторные и структурные функции. Кроме того, в геноме эукариот часто встречаются участки, принадлежащие «генетическим паразитам», например, транспозонам.
Кодирование начинается с того, что все буквы, цифры и изображения переводят в двоичный код, то есть последовательность нулей и единиц, а их уже — в последовательность нуклеотидов, то есть четверичный код.
Считывать ДНК можно по-разному. Самая распространенная методика — цепочку молекулы ДНК копируют с помощью оснований, у каждого из которых есть цветовая метка. Затем очень чувствительный детектор считывает данные, и по цветам компьютер восстанавливает последовательность нуклеотидов.
Как в ДНК появляется новая информация
Делается это при помощи технологии CRISPR-Cas9, ее еще называют генетическими ножницами. Она была разработана восемь лет назад, а в 2020 году удостоена Нобелевской премии по химии.
Ранее записывать информацию нужно было долго и при помощи специального оборудования. Однако группа ученых из Колумбийского университета автоматизировала этот процесс.
Нам удалось научить клетки разговаривать с компьютером посредством электронных сигналов и таким образом скачивать информацию с любого электронного носителя.
Харрис Ванг, профессор системной биологии
Авторы объясняют, что они переводят двоичный код компьютерной программы в электрические импульсы, которые посылают в клетку. На ее поверхности есть рецепторы, которые воспринимают эти сигналы и уже переводят их на язык ДНК, автоматически выстраивая нужную последовательность генома.
В результате к цепочке ДНК добавляется так называемый прицеп, или дополнительный фрагмент. В отличие от цифровой компьютерной информации, он представляет собой набор букв генетического кода, то есть аналоговый шифр, поэтому ученый сравнивает этот отрезок с магнитной лентой.
Какой объем информации можно записать в ДНК
С помощью новой технологии сотрудников Колумбийского университета удалось закодировать и прочитать 2,14 МБ информации. Итоговая физическая плотность записи составила 215 000 000 ГБ на грамм нуклеиновой кислоты.
Один оборот спирали ДНК в B-форме — это примерно 10 пар нуклеотидов. Кодирующей будет одна из нитей, так как вторая всегда комплиментарна первой.
Таким образом, есть 10 ячеек, в каждой из которых может быть одна из четырех букв: А, Т, Г, Ц.
При использовании четвертичного или двоичного кодирования плотность кодирования информации в ДНК составляет два бита на ячейку, то есть 20 бит на один оборот спирали, линейный размер которого примерно 3,4 нм объемом
11 м 3 — это то, что можно записать.
Сегодня можно создавать процессоры, в которых 1 бит записывается на 10 нанометрах. Таким образом, в ДНК, исходя из линейных размеров, можно записать примерно в 60 раз больше информации.
Насколько надежно записывать информацию на ДНК
В марте 2017 года журнал Science опубликовал статью американских ученых, которым удалось записать 2*10 17 байт на грамм ДНК. Биологи подчеркивают, что не потеряли ни байта.
К несомненным преимуществам записи информации на ДНК относится огромная плотность хранения данных, а также стабильность носителя — правда, лишь при низких температурах.
В ДНК информация записана в трехмерном аналоговом виде, а это наиболее устойчивая форма. В таком виде данные могут храниться сотни тысяч, а то и миллионы лет, заявил профессор системной биологии Харрис Ванг.
Вывод
Несмотря на все преимущества, технология записи информации на ДНК находится на начальном этапе своего развития. На сегодняшний день синтез ДНК остается все еще очень дорогим, поэтому за мегабайт данных, записанных на ДНК-«флешку», придется заплатить порядка 3,5 тыс. долларов.
Ученым еще предстоит разработать технологию автоматической передачи информации с ДНК. Также важно упростить способ передачи информации из компьютера в клетку. Сейчас для этого используется поток электронов, но в будущем его заменят чем-нибудь другим.
Например, переменным магнитным полем или температурой внешней среды. Или даже обычным лучом света — ведь фоторецепторы есть у большинства живых организмов.
Вирусы: назад к истокам
Вероятно, уже в недалеком будущем информацию будут хранить в ДНК, а термин «вирус» в данном контексте получит буквальное значение.
Помните ли вы исходное значение слова «вирус»? Да-да, я имею в виду тот самый биологический объект, в честь которого получили свое название зловредные компьютерные программы, помещающие свой код внутрь других файлов с целью воспроизводства и распространения.
Вполне вероятно, что в обозримом будущем значение этого слова применительно к компьютерным данным получит свое оригинальное значение. Дело в том, что этим летом исследователям из Microsoft и Университета Вашингтона удалось сделать то, что не удавалось сделать до них никому, — записать 200 Мбайт данных в виде последовательности нуклеотидов, входящих в состав искусственно созданной ДНК.
Какое отношение к этому имеют вирусы? Да самое прямое! Вирусы внедряют свой генетический код в ДНК клеток пораженных организмов, заставляя их воспроизводить себя, а не полезные для организма белки (напомню, что жизнь, как учили нас классики, — это форма существования белковых тел).
Особенно агрессивные вирусы настолько мешают нормальной работе пораженного ими организма, что в итоге приводят к его смерти. Точно так же особенно неприятный вредоносный код может привести к невозможности использовать пораженную информационную систему.
Поэтому, раз уж человечество начинает активно записывать информацию в виде ДНК, пожалуй, стоит задуматься о защите информации на «аппаратном уровне». Для начала расскажем вам, как устроено «железо», с которым нам предстоит иметь дело.
Как устроена ДНК
ДНК, или дезоксирибонуклеиновая кислота, — это носитель генетической информации и по совместительству — самая большая молекула в нашем организме. Если использовать аналогии из сферы информационных технологий, это такой загрузочный образ операционной системы. На основании ДНК синтезируются РНК — рибонуклеиновые кислоты, играющие роль программ для синтеза белков («исполняемых модулей» в компьютерных терминах), из которых и состоят все живые организмы и которые отвечают за протекание физиологических процессов на молекулярном уровне.
Все признаки организма, начиная от цвета волос и глаз и заканчивая предрасположенностью к наследственным заболеваниям, записаны в ДНК. Записаны они в виде последовательности нуклеотидов — молекулярных блоков, содержащих в себе всего лишь четыре разновидности азотистых оснований: аденин, гуанин, тимин, цитозин. Это такие биологические биты.
Как видите, в отличие от человека, матушка-природа использовала не двоичную систему счисления, а четверичную. Кстати, природа хорошо позаботилась о защите от сбоев — у большинства живых существ ДНК представляет собой не одну, а две цепочки нуклеотидов, закрученные друг вокруг друга как витая пара в двойную спираль.
Держатся эти две цепочки друг за друга водородными связями, которые образуются только в том случае, если с каждой из сторон расположен строго определенный нуклеотид, — таким образом автоматически гарантируется взаимное соответствие информации в каждой из двух спиралей. На этом и основан первый механизм защиты от сбоев: при расшифровке или репликации ДНК используется одна из двух спиралей, а вторая играет роль контрольной — на тот случай, если вдруг какая-то последовательность нуклеотидов, кодирующих тот или иной генетический признак, оказалась в одной из спиралей повреждена.
Кроме взаимного соответствия двух цепочек нуклеотидов кодирование наследственных признаков дополнительно производится с применением избыточного алгоритма — можно сказать, что каждый наследственный признак, записанный в виде последовательности биологических битов — оснований, дополнительно снабжен контрольной суммой.
За те полвека, что прошли с момента открытия ДНК, эти последовательности довольно неплохо изучены, что позволяет любому желающему заказать расшифровку основных генетических признаков собственной ДНК онлайн, причем не только в ближайшей лаборатории, но и в Интернете — с помощью сервиса 23andme и аналогичных ему.
Как считывают ДНК
Теперь о том, как информацию ДНК считывают. Изначально в распоряжении ученых были такие методы, как рентгеновский структурный анализ, семейство спектроскопических методов и масс-спектрометрия. Все эти методы неплохо работают для небольших молекул, состоящих из двух, трех, четырех атомов, но все становится сильно сложнее, когда количество атомов действительно велико.
Однако ДНК не зря считают самой большой молекулой в нашем организме — в человеческой ДНК из гаплоидной клетки содержится порядка 3 млрд пар оснований. Ее молекулярная масса на несколько порядков больше молекулярной массы самого крупного из известных науке белков.
В общем, это неимоверно огромная куча атомов, поэтому на расшифровку данных при использовании классических методов считывания даже сегодня, с применением суперкомпьютеров, легко уходят месяцы, а то и годы.
Но ученым удалось придумать метод секвенирования, который сильно ускоряет процедуру. Основная его идея — разбиение одной длинной последовательности атомов на много коротких фрагментов, которые можно анализировать параллельно, тем самым кратно увеличивая скорость расшифровки.
Для секвенирования биологи используют «молекулярные машины» — специальные белки (энзимы) полимеразы. Основная функция этих белков — копирование ДНК. Делают они это, последовательно проходя вдоль спирали и собирая из нуклеотидов идентичную молекулу.
Но поскольку нам нужна не просто полная копия ДНК, а нарезка на короткие фрагменты, то дополнительно используют так называемые праймеры и маркеры — соединения, сообщающие полимеразе, где начать клонировать, а где закончить.
Праймеры представляют собой четко определенную последовательность нуклеотидов, которая присоединяется к цепочке лишь там, где встречает «ответную» комбинацию. Полимераза находит праймер, «садится» на цепочку нуклеотидов и начинает достраивать ее из компонент, которые помещены в раствор. И делает это до тех пор, пока не встретит маркер — модифицированный нуклеотид, на котором дальнейшая «достройка» цепочки обрывается.
Определенную проблему представляет тот факт, что в рамках этого метода невозможно указать точные «адреса» начала и конца клонирования, а указать можно лишь те последовательности «битов», с которых начинается и которыми заканчивается выделение фрагмента.
Создан язык программирования клеток с помощью молекул ДНК http://t.co/AgiFAcy5o8 #habr
Если говорить в компьютерных терминах, то происходит это следующим образом. Допустим, у нас есть комбинация бит 1101100001010111010010111. Предположим, что нашим праймером является комбинация 0000, а маркером — комбинация 11. В результате секвенирования мы получим следующий набор фрагментов, в порядке убывания их вероятности: 0000101011, 00001010111, 0000101011101001011, 00001010111010010111.
Варьируя праймер и маркер, мы в конечном итоге переберем все возможные комбинации бит, считаем их, а после считывания восстановим из отдельных фрагментов всю последовательность.
Выглядит немного сложно и неочевидно, но это действительно работает и обеспечивает неплохую скорость, поскольку в итоге все необходимые действия можно делать параллельно. Неплохая скорость по меркам биологов — это несколько часов. Существенно лучше вышеупомянутых месяцев или даже лет, но по меркам ИТ, скажем так, многовато.
ДНК и хранение произвольной информации
Научившись за полвека неплохо считывать информацию из ДНК, оставалось научиться синтезировать цепочки нуклеотидов. Тут надо уточнить, что исследователи Microsoft были не первыми, кто записал информацию в виде двойной спирали ДНК. Первыми были ученые из европейского института биоинформатики (EMBL-EBI), несколько лет назад записавшие 739 Кбайт.
«ДНК-накопитель»: https://t.co/p5hGLRuJDC. Microsoft удалось записать 200 МБ данных на нитях синтетической ДНК. pic.twitter.com/RZkMyMEBEC
В чем же новизна достижений Microsoft? Во-первых, в существенном увеличении объема записи — до 200 Мбайт. Уже довольно близко к тем 750 Мбайт, которые содержатся в ДНК человека. Впрочем, главная инновация состоит в том, что исследователи предложили способ, позволяющий считывать не всю ДНК целиком, а ее отдельный участок — порядка 100 битов-оснований за одну операцию.
А добились они этого путем использования таких пар праймеров и маркеров, которые обеспечивают копирование полимеразой — и последующее считывание — блока данных строго определенного размера, расположенного по определенному адресу относительно начала «файла» — цепочки нуклеотидов. Это все еще не совсем полный аналог произвольного доступа к памяти, но довольно близкое к нему поблочное чтение.
Пока ученые считают, что основной нишей подобного использования ДНК могут стать модули памяти высокой плотности, предназначенные для длительного хранения информации. В этом есть смысл — плотность записи данных в лучших современных образцах флеш-памяти достигает десятков квадриллионов (
10 16 ) бит на кубический сантиметр, в то время как плотность хранения данных в ДНК на три порядка выше: десятки квинтиллионов (
Дополнительное преимущество состоит в том, что молекулы ДНК достаточно стабильны и, с учетом алгоритмов коррекции ошибок, позволяют хранить информацию годами, а то и веками.
Microsoft закупила 10 млн нитей синтетической ДНК: https://t.co/d93pe8IWQn. Будут экспериментировать с долговременным хранением данных.
Вернемся к вирусам
Что это означает с точки зрения информационной безопасности? А означает это, что целостности записанной в таком видео информации угрожают организмы, которые специализируются на порче данных уже миллиарды лет, — вирусы.
Конечно, ожидать появления специальных генно-модифицированных вирусов, заточенных «охотиться» именно на подобные ДНК, в которые записана какая-то информация, не стоит. Просто потому, что модифицировать данные, внедряя в них вредоносный код, проще, пока эти данные представлены в чисто цифровом виде — еще до записи в ДНК.
А вот надо ли будет думать о защите от обычных вирусов, работая с таким запоминающим устройством, — вопрос открытый. Ведь если в раствор с ДНК попадет, например, вирус насморка, полимераза, скорее всего, будет реплицировать и его тоже.
Поэтому как бы не пришлось, прочитав ДНК-чип лет через десять после его записи, вспоминать, не чихала ли лаборантка во время записи важного архивного документа.